アンプリコンシーケンスデータ解析 (QIIME2)
特長
-
Amplicon Sequence Variant (ASV) 法により、相同率の高い近縁種同士の識別が可能
-
学術論文で求められる二次解析 (多様性解析や統計解析の機能) が充実
概要
QIIME21)は代表配列の決定から多様性解析まで、アンプリコンシーケンス解析に必要なソフトウェアをまとめたオープンソースの解析パイプラインです。解析結果はqzvという独自のファイル形式になり、専用のwebサイトQIIME 2 View2)で閲覧でき、前身のQIIME13)と比べデータのアウトプットにおいてGUI的な側面も強化されています。
QIIME2がQIIME1と大きく異なる点は、代表配列の決定方法にあります。QIIME1で用いられるOperational Taxonomic Unit (OTU) 化は、一定の相同率 (例えば97%以上) の配列同士を同種由来とみなして代表配列をまとめていく方法です。
QIIME2のASV法は、数塩基の差異しかない配列同士について、その差異がPCRやシーケンシングの過程で生じたエラーに起因するのか、種内変異等の生物学的なイベントによるものなのかを統計学的に区別する代表配列決定法です。
これにより、今まで「相同率97%だが実は別種であった可能性のある配列」を区別することで、より解像度の高いデータ解析を可能としています。
参考文献
-
(1)
Bolyen E, Rideout JR, Dillon MR, Bokulich NA, Abnet CC, et al. Reproducible, interactive, scalable and
extensible microbiome data science using QIIME 2. Nat Biotechnol 2019;37:852–857. -
(3)
Caporaso JG, Kuczynski J, Stombaugh J, Bittinger K, Bushman FD, et al. QIIME allows analysis of high-throughput community sequencing data. Nat Methods 2010;7:335–336.
解析の流れ
-
fastqデータSample Typeの入力
-
プライマー配列の除去
-
代表配列の決定(ASV法)
-
相同性検索
-
多様性解析
-
統計解析
仕様・納品内容
報告サンプル
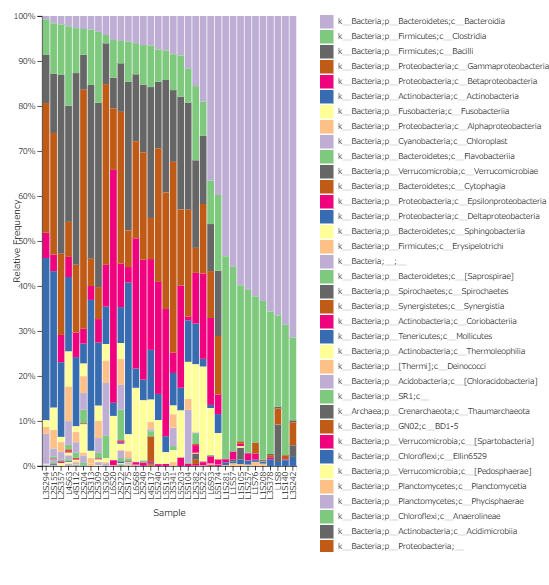
相同性検索結果 ( 表、 バーチャート )
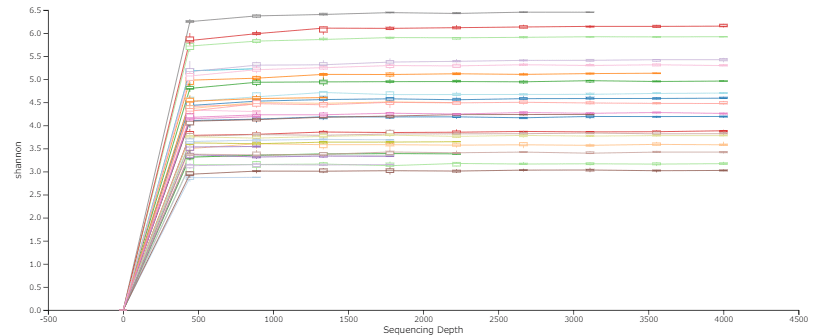
αレアファクションカーブ
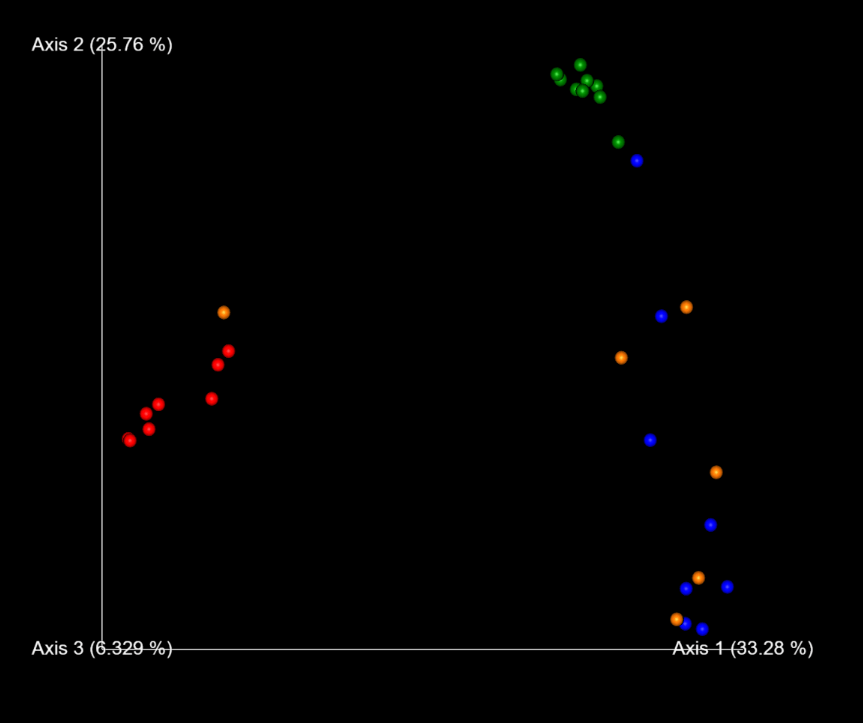
3D 主座標分析
価格・納期
試験項目 | 検体数 | 単位 | 単価 (税抜) | 納期 |
アンプリコンシーケンスデータ解析 (QIIME2) 一次解析 | 3 ~ | 式 | 30,000 円 | 10 営業日 |
アンプリコンシーケンスデータ解析 (QIIME2) 二次解析 | 3 ~ | 式 | 30,000 円 | 10 営業日 |
アンプリコンシーケンスデータ解析 (QIIME2) 一次解析~二次解析 | 3 ~ | 式 | 60,000 円 | 10 営業日 |
- 遺伝子機能の予測解析をご希望の場合には、「PICRUSt2による予測メタゲノム解析」をご参照下さい。
ご依頼前の同意事項
・ メディア(DVD-R)による配送納品のみとなります。
・ ご依頼前の同意事項 (共通) を必ずご確認下さい。
・ ご依頼前の解析手法などの選択についてのご相談には可能な範囲で対応しますが、最終的なご判断はお客様の責任にてお願いいたします。
