PICRUSt2による予測メタゲノム解析
特長
-
16S rDNAのアンプリコンシーケンス解析データ (QIIME2) から、 遺伝子の機能を予測
-
予測された遺伝子に付与された2種類の機能分類に基づく遺伝子組成を推定
・Clusters of Orthologous Genes (COG):COGデータベースに基づく機能分類
・Enzyme Commision numbers (EC):酵素の種類に基づいた機能分類
概要
参考文献
-
(1)
Douglas GM, Maff ei VJ. Zaneveld JR, Yurgel SN, Brown JR, et al. PICRUSt2 for prediction of metagenome
functions. Nat. Biotechnol. 2020 ;38(6):685–688
受入可能な検体
検体の種類 | 形式 |
QIIME2 ( 一次解析 ) 出力データファイル | qza, qzv ファイル |
- アンプリコンシーケンス解析の生データでご依頼の場合は、QIIME2 の一次解析が必要です。
解析の流れ
-
代表配列をリファレンス配列へアライメント
-
系統樹の作成
-
代表配列リード数のノーマライズ
-
COG、ECを使用した遺伝子の機能予測
-
遺伝子の機能組成表の集計
仕様
解析ソフトウェア | 解析データベース | 内容 |
PICRUSt2 | Clusters of Orthologous Genes (COG) および Enzyme Commision numbers (EC) | QIIME2 で出力した代表配列とリード数を基に、細菌叢が持つ遺伝子の機能を予測します。 |
納品内容
報告内容 | 形式 | |
系統樹 | リファレンス配列と検体の代表配列を含めて描画した系統樹 | tre ファイル |
補正された代表配列の存在量 | リファレンス配列のゲノム情報に含まれる 16S rDNAコピー数を基に補正された、 代表配列の存在量 | tsv ファイル |
Nearest-sequenced taxon index | 遺伝子機能の予測精度の評価結果 | |
予測された遺伝子の存在量 | 2 種類のデータベース (COG、 EC) を用いた遺伝子機能予測の結果と、 各遺伝子の推定存在量 | |
報告サンプル
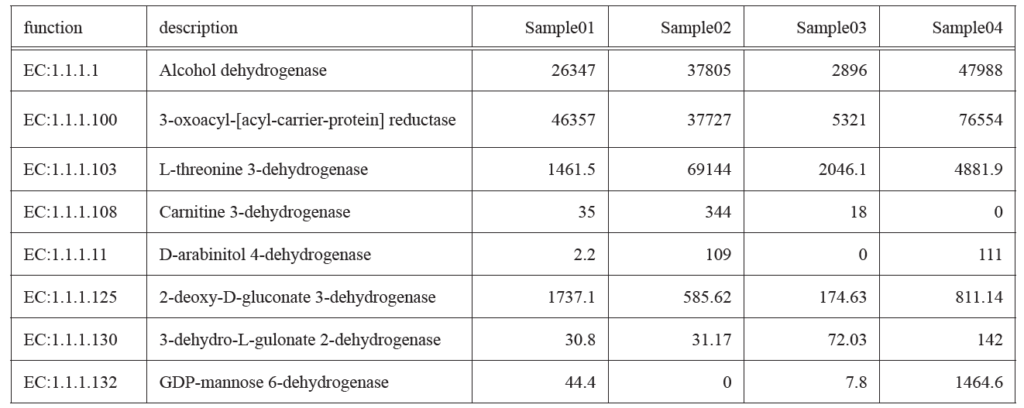
価格・納期
試験項目 | 検体数 | 単位 | 単価(税抜) | 目安納期 |
PICRUSt2 による予測メタゲノム解析※ | 1 ~ | 式 | 28,000 円 | 8 営業日 |
※ QIIME2の出力データ(一次解析結果)が必須となります。 当社仕様で実施していないQIIME2の出力データの場合はお問い合わせ下さい。
ご依頼前の同意事項
・ メディア(DVD-R)による配送納品のみとなります。
・ ご依頼前の同意事項(共通)を必ずご確認下さい。
・ ご依頼前の解析手法などの選択についてのご相談には可能な範囲で対応しますが、最終的なご判断はお客様の責任にてお願いいたします。
