Metagenome@KINソフトウェアをバージョンアップしました。
ソフトウェアをバージョンアップしました。
Metagenome@KINソフトウェアをVer.2.21からVer.2.3に更新したことで、以下の解析可能になりました。
分類階級 (界から種レベル) ごとに
α多様性指数を出力できるようになりました。
Simpson’s Index, Shannon Entropyを出力します。微生物群集の多様性を考察できるようになりました。

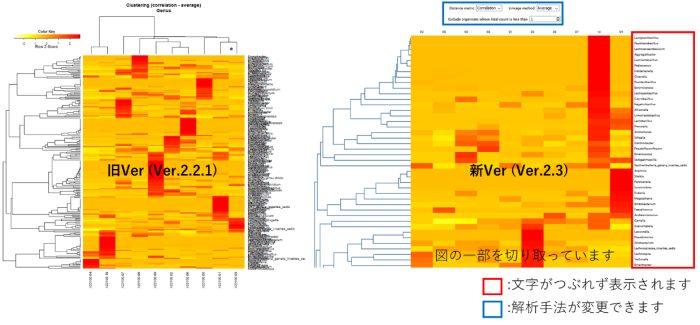
ヒートマップ・クラスター分析結果の
仕様が変更されます。
これまでpng形式の画像のみのご報告でしたが、html上で距離行列および系統解析手法がご自身で選択できるようになり、クラスタリングに使用する菌のリード数の下限値も設定できます。また、旧バージョンに比べて、解像度の高い図を出力できるようになりました。
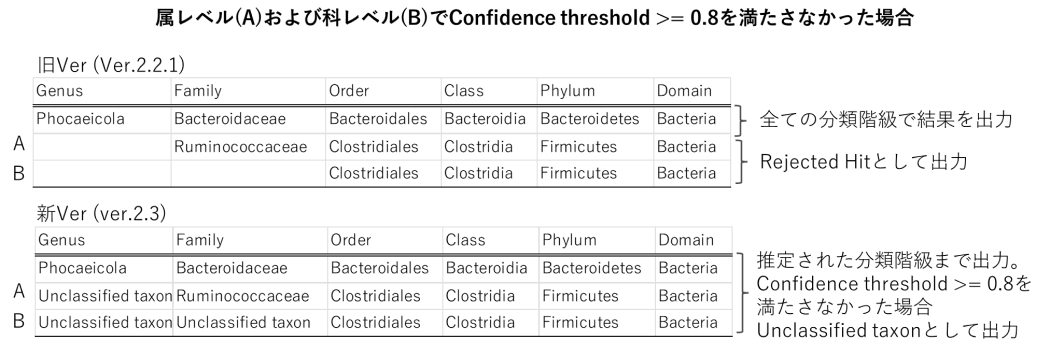
「Rejected_hit (分類群を同定できない) 」が「Unclassified hit」および「Unclassified taxon」に変更されます
【RDP、テクノスルガ・ラボ微生物同定DB共通】
各データベースで表1の条件を満たさなかった配列は「Unclassified hit」として出力されます。
【RDPのみ】
これまで、界~属レベルのいずれかでConfidence threshold >= 0.8を満たさなかった場合、その配列は「Rejected_hit」として出力されていました。一方でソフトウェアのバージョンアップにより、例えば、属レベルでConfidence threshold >= 0.8を満たさなかった場合、界から科レベルまでは帰属分類群名が同定され、属レベルでは「Unclassified taxon」となります。
今後のデータ解析について
機能が追加されたアンプリコンシーケンス解析をぜひご利用ください。すでに「Ver.2.21」で解析済みのデータを「Ver.2.3」で改めて解析することも可能です (再解析費用が発生します) 。「Ver.2.2.1」でのデータ解析も引き続き、ご指定いただくことで承ります。
試験に関するお問い合わせ
株式会社テクノスルガ・ラボ 営業部
E-mail:tsl-contact@tecsrg.co.jp
電話番号:054-349-6211
